忘記密碼
實驗室計算生物團隊新突破:通過側鏈建模研究蛋白質突變的影響及序列改造
近日,上海人工智能實驗室聯合復旦大學提出蛋白質點突變建模新方法——通過側鏈建模研究蛋白質突變的影響及序列改造。該方法由上海人工智能實驗室計算生物學創新中心教授、復旦大學復雜體系多尺度研究院院長馬劍鵬和諾貝爾化學獎得主Michael Levitt教授共同帶領的科研團隊提出。團隊從蛋白質側鏈結構出發對點突變問題進行深入探索,并利用早前開發的新算法(OPUS-Mut)指導蛋白質設計,為研究蛋白質突變和蛋白質設計問題提供了新的利器。目前,該成果已開源,相關論文“OPUS-Mut: Studying the Effect of Protein Mutatio through Side-Chain Modeling”在線發表于知名國際期刊Journal of Chemical Theory and Computation。
蛋白質點突變在蛋白質工程和蛋白質進化的研究中起重要作用,也是諸多重大遺傳疾病的發病原因,因此,對點突變的結構進行建模意義重大。然而,目前最優的蛋白質結構預測算法AlphaFold2并不適合直接應用于點突變建模[1],這使得高精度點突變結構建模成為全球計算生物界普遍面臨的難題。此次團隊提出的通過側鏈建模研究蛋白質突變影響的方法,正是針對這一世界難題做出的突破性研究。
從結構角度來說,蛋白質點突變可以近似地看作是相應殘基的側鏈替換。因此,從側鏈結構出發對點突變問題進行研究是一條可行之路。經過算法改進和模型設計,團隊所開發的算法對突變位點側鏈構象的預測有著較高的靈敏度(圖1)。同時,通過對比野生蛋白和突變蛋白的側鏈建模結果,可以得到由突變引起的、對蛋白中其它殘基的側鏈結構擾動數據,進而預測該突變對蛋白功能的影響。研究團隊收集了293個T4溶菌酶點突變對穩定性的影響數據,并將其分為提升穩定性組與降低穩定性組。結果顯示(圖2),兩組的突變對突變前后其他殘基側鏈擾動的大小存在顯著性差異,且降低穩定性組的突變對其他殘基擾動更大——在293個突變中,擾動最大的前10個突變中有9個屬于降低穩定性組。與此同時,該篇論文中的其他實驗也得出了類似結果,這些實驗共同驗證了新算法可以用來區分“溫和型突變”和“破壞型突變”,即對目標蛋白中其他殘基的側鏈結構擾動越大,則該突變更可能為“破壞型突變”。由于該算法并未使用任何突變-生物功能變化數據進行訓練,故不存在過擬合現象,可以和相關模型形成良好互補。

圖1. 新算法對野生型肌紅蛋白及其突變體在突變位點的側鏈建模結果。a)為突變位點的周圍環境。b-f)分別為野生型肌紅蛋白和其不同突變體的側鏈建模結果。其中紅色為突變位點真實實驗結構,藍色為突變位點周圍殘基的真實實驗結構,黃色為預測結果。

圖2. 提升穩定性組和降低穩定性組的側鏈擾動大小(Sdiff)。顯著性分析采用Mann-Whitney U-test。
此外,研究團隊以T4溶菌酶為目標蛋白,遍歷其所有可能的點突變(164×19),之后計算不同點突變對其它殘基側鏈的擾動情況,并對其按照擾動大小進行排序,然后選用對其它殘基側鏈擾動最小的那部分點突變進行組合,得到了一個具有60%序列相似度的T4溶菌酶的突變體(圖3)。同時,在不采用模板信息的情況下使用AlphaFold2對該突變體進行結構預測,預測結果與原始野生型蛋白的真實實驗結構的相似度指標TM-score為0.95。該結果顯示,新算法可以在降低序列同源性的同時,保持目標蛋白的生物學功能,從而豐富具有相同功能的蛋白序列多樣性,助力蛋白質設計。值得注意的是,由于其并未用到多序列比對信息,故可以和基于多序列比對及序列采樣信息的蛋白質幻想設計方法形成良好互補,對其采樣提供一定指導。
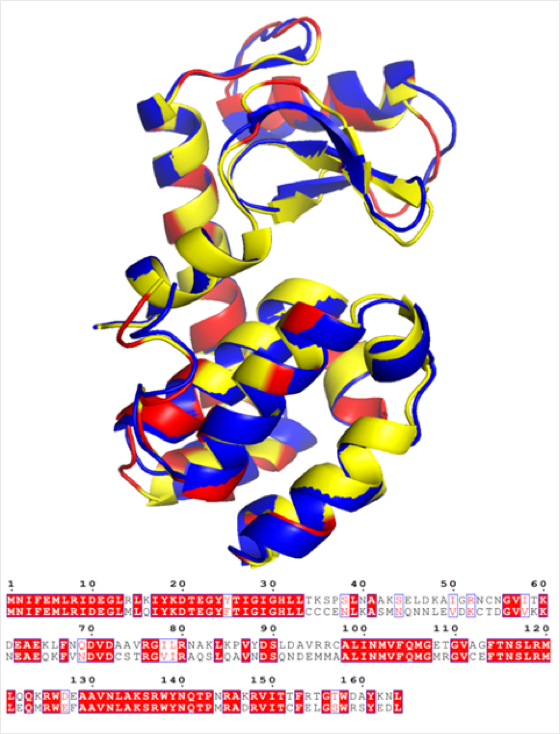
圖3. 具有60%序列相似度的T4溶菌酶突變體的AlphaFold2預測結果。藍色為原始野生型蛋白的真實實驗結構,黃色為AlphaFold2對突變體的預測結構,紅色為突變位點。
馬劍鵬教授與Michael Levitt教授的聯合團隊在蛋白質結構預測和設計領域深耕已久,科研成果發表于Nature、Science、PNAS等國際頂刊。近年來,團隊相繼開發了OPUS-Fold、OPUS-Refine、OPUS-X、OPUS-Rota4及OPUS-Mut等一系列算法,可分別應用于不同場景,為蛋白質結構預測和設計提供了堅實的技術保障。尤其是對基于蛋白質大分子藥物的設計與優化,團隊具有干濕結合、AI賦能的全鏈條一體化的研發優勢。
上海人工智能實驗室計算生物學創新中心以人工智能為技術中樞,構建新一代生物體系分析工具與方法,解讀生物遺傳信息,加速生命科學在分子層面的研究。通過對蛋白質、DNA等分子功能結構的預測與設計,打破生物數據壁壘,為生命科學研究提供新思路、新方法。
[1] G. R. Buel, K. J. Walters, Can AlphaFold2 predict the impact of missense mutations on structure? Nat Struct Mol Biol29, 1-2 (2022).


